よむ、つかう、まなぶ。
資料3-2 鈴木先生提出資料 (73 ページ)
出典
| 公開元URL | https://www.mhlw.go.jp/stf/seisakunitsuite/bunya/0000121431_00348.html |
| 出典情報 | 新型コロナウイルス感染症対策アドバイザリーボード(第89回 6/30)《厚生労働省》 |
ページ画像
ダウンロードした画像を利用する際は「出典情報」を明記してください。
低解像度画像をダウンロード
プレーンテキスト
資料テキストはコンピュータによる自動処理で生成されており、完全に資料と一致しない場合があります。
テキストをコピーしてご利用いただく際は資料と付け合わせてご確認ください。
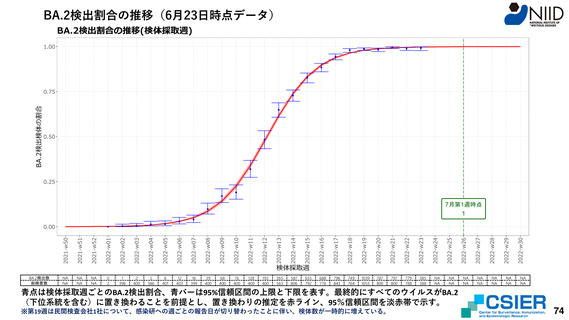
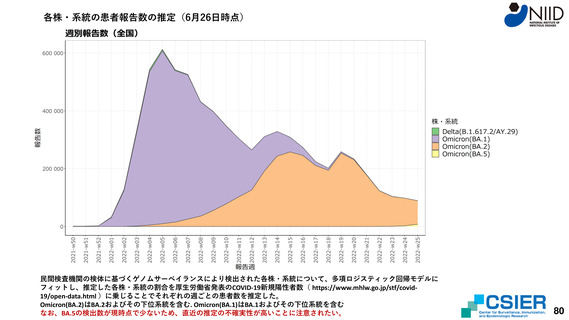
民間検査機関の検体に基づくゲノムサーベイランス(検証中)によるBA.2, BA.5検出の推定
背景
全国の変異株の発生動向を監視するためのゲノムサーベイランスの確立を目指し、今般、民間検査機関から得られた全国800(第12週までは400)検体を用いた検証を感染
研で行うこととした。
対象
•
国内の民間検査機関2社に集められた検体
•
全国(※)で合計800検体/週を目途に検査(A社400検体/週、B社検体400/週)
•
毎日、検査機関側でA社では57(火曜日~土曜日)~115(月曜日)検体、B社では65~70(平日)、~40(土曜日)検体を抽出した後、ゲノム解析検査を実施し、感染
研病原体ゲノム解析研究センターのCOG-Jpを用いたデータ解析後に、週ごとに感染研病原体ゲノム解析研究センターに報告(同時に感染研病原体ゲノム解析研究セン
ターでもCOG-Jpで共有されたデータを解析)
※ A社では、全国一律の検体プールからランダムに抽出。B社では、10のエリアに分けた地域ごとにサンプル数を決め、地域ごとにランダムに抽出。地域性を一定程度
考慮しているが、分布については検討中。
BA.2およびBA.5検出率解析方法
•
ゲノム解析データを基に、PANGO lineageを決定(病原体ゲノム解析研究センターで実施)。
•
最終的に全てのウイルスがオミクロン株BA.2、BA.5に置き換わることを前提に、Lineageが判明した検体数(解析不能分を除く)に占めるBA.2、BA.5検出検体の割合につ
いて、ロジスティック成長モデルにフィットさせ推定を行った。また、各系統・株の検出割合について、多項ロジスティック回帰モデルにフィットさせ推定を行った。
特徴
•
都道府県別のランダムな対象の抽出に厳密な基準を設定していないこと、及び各地域の対象数を考慮すると、地域(都道府県別)の偏りについては検査時点では考慮不
可(後に判明)であり、地域ごとの代表性の確保はできない(原則、全国と限られた地域での分析のみ考慮)。
•
本サーベランスの対象は、民間検査機関に集められた検体で、個別に医療機関を受診した症例の検査検体が中心であり、集団発生の影響が比較的少なく、実際の地域の
感染状況を反映しやすいと考えられる。
•
GISAIDのデータより、1~2週間早く解析できる。
•
今後、実際のBA.2、BA.5検出の推移と本推定との検証が必要。
検証の中間評価
•
検査会社により検体の抽出方法は異なるが、全国一律の検体プールからランダムに抽出するA社に限定した場合でも全国的な傾向は同様であった。
73
背景
全国の変異株の発生動向を監視するためのゲノムサーベイランスの確立を目指し、今般、民間検査機関から得られた全国800(第12週までは400)検体を用いた検証を感染
研で行うこととした。
対象
•
国内の民間検査機関2社に集められた検体
•
全国(※)で合計800検体/週を目途に検査(A社400検体/週、B社検体400/週)
•
毎日、検査機関側でA社では57(火曜日~土曜日)~115(月曜日)検体、B社では65~70(平日)、~40(土曜日)検体を抽出した後、ゲノム解析検査を実施し、感染
研病原体ゲノム解析研究センターのCOG-Jpを用いたデータ解析後に、週ごとに感染研病原体ゲノム解析研究センターに報告(同時に感染研病原体ゲノム解析研究セン
ターでもCOG-Jpで共有されたデータを解析)
※ A社では、全国一律の検体プールからランダムに抽出。B社では、10のエリアに分けた地域ごとにサンプル数を決め、地域ごとにランダムに抽出。地域性を一定程度
考慮しているが、分布については検討中。
BA.2およびBA.5検出率解析方法
•
ゲノム解析データを基に、PANGO lineageを決定(病原体ゲノム解析研究センターで実施)。
•
最終的に全てのウイルスがオミクロン株BA.2、BA.5に置き換わることを前提に、Lineageが判明した検体数(解析不能分を除く)に占めるBA.2、BA.5検出検体の割合につ
いて、ロジスティック成長モデルにフィットさせ推定を行った。また、各系統・株の検出割合について、多項ロジスティック回帰モデルにフィットさせ推定を行った。
特徴
•
都道府県別のランダムな対象の抽出に厳密な基準を設定していないこと、及び各地域の対象数を考慮すると、地域(都道府県別)の偏りについては検査時点では考慮不
可(後に判明)であり、地域ごとの代表性の確保はできない(原則、全国と限られた地域での分析のみ考慮)。
•
本サーベランスの対象は、民間検査機関に集められた検体で、個別に医療機関を受診した症例の検査検体が中心であり、集団発生の影響が比較的少なく、実際の地域の
感染状況を反映しやすいと考えられる。
•
GISAIDのデータより、1~2週間早く解析できる。
•
今後、実際のBA.2、BA.5検出の推移と本推定との検証が必要。
検証の中間評価
•
検査会社により検体の抽出方法は異なるが、全国一律の検体プールからランダムに抽出するA社に限定した場合でも全国的な傾向は同様であった。
73