よむ、つかう、まなぶ。
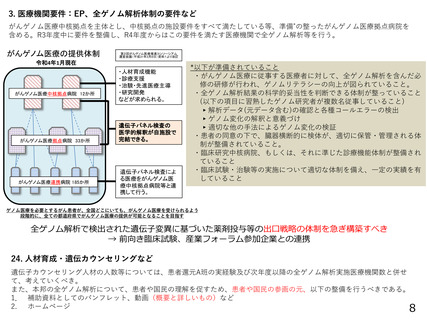
資料3-1 「全ゲノム解析等に係る厚生労働科学研究班」からの報告 (10 ページ)
出典
| 公開元URL | https://www.mhlw.go.jp/stf/newpage_23993.html |
| 出典情報 | 厚生科学審議会 科学技術部会全ゲノム解析等の推進に関する専門委員会(第8回 3/2)《厚生労働省》 |
ページ画像
ダウンロードした画像を利用する際は「出典情報」を明記してください。
低解像度画像をダウンロード
プレーンテキスト
資料テキストはコンピュータによる自動処理で生成されており、完全に資料と一致しない場合があります。
テキストをコピーしてご利用いただく際は資料と付け合わせてご確認ください。
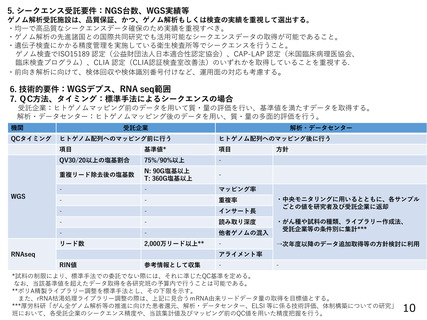
5. シークエンス受託要件:NGS台数、WGS実績等
ゲノム解析受託施設は、品質保証、かつ、ゲノム解析もしくは検査の実績を重視して選出する。
・均一で高品質なシークエンスデータ確保のため実績を重視すべき。
・ゲノム解析の先進諸国との国際共同研究でも活用可能なシークエンスデータの取得が可能であること。
・遺伝子検査にかかる精度管理を実施している衛生検査所等でシークエンスを行うこと。
ゲノム検査でISO15189 認定(公益財団法人日本適合性認定協会)、CAP-LAP 認定(米国臨床病理医協会、
臨床検査プログラム)、CLIA 認定(CLIA認証検査室改善法)のいずれかを取得していることを重視する.
・前向き解析に向けて、検体回収や検体識別番号付けなど、運用面の対応も考慮する。
6. 技術的要件:WGSデプス、RNA seq範囲
7. QC方法、タイミング:標準手法によるシークエンスの場合
受託企業:ヒトゲノムマッピング前のデータを用いて質・量の評価を行い、基準値を満たすデータを取得する。
解析・データセンター:ヒトゲノムマッピング後のデータを用い、質・量の多面的評価を行う。
機関
QCタイミング
WGS
受託企業
解析・データセンター
ヒトゲノム配列へのマッピング前に行う
ヒトゲノム配列へのマッピング後に行う
項目
基準値*
項目
QV30/20以上の塩基割合
75%/90%以上
-
重複リード除去後の塩基数
N: 90G塩基以上
T: 360G塩基以上
-
-
-
マッピング率
-
-
重複率
-
-
インサート長
-
-
読み取り深度
-
-
他者ゲノムの混入
リード数
2,000万リード以上**
-
RNAseq
方針
・中央モニタリングに用いるとともに、各サンプル
ごとの値を研究者及び受託企業に返却
・がん種や試料の種類、ライブラリー作成法、
受託企業等の条件別に集計***
→次年度以降のデータ追加取得等の方針検討に利用
アライメント率
RIN値
参考情報として収集
-
-
*試料の制限により、標準手法での委託でない際には、それに準じたQC基準を定める。
なお、当該基準値を超えたデータ取得を各研究班の予算内で行うことは可能である。
**ポリA精製ライブラリー調整を標準手法とし、その下限を示す。
また、rRNA枯渇処理ライブラリー調整の際は、上記に見合うmRNA由来リードデータ量の取得を目標値とする。
***厚労科研「がん全ゲノム解析等の推進に向けた患者還元、解析・データセンター、ELSI 等に係る技術評価、体制構築についての研究」
班において、各受託企業のシークエンス精度や、当該集計値及びマッピング前のQC値を用いた精度把握を行う。
10
ゲノム解析受託施設は、品質保証、かつ、ゲノム解析もしくは検査の実績を重視して選出する。
・均一で高品質なシークエンスデータ確保のため実績を重視すべき。
・ゲノム解析の先進諸国との国際共同研究でも活用可能なシークエンスデータの取得が可能であること。
・遺伝子検査にかかる精度管理を実施している衛生検査所等でシークエンスを行うこと。
ゲノム検査でISO15189 認定(公益財団法人日本適合性認定協会)、CAP-LAP 認定(米国臨床病理医協会、
臨床検査プログラム)、CLIA 認定(CLIA認証検査室改善法)のいずれかを取得していることを重視する.
・前向き解析に向けて、検体回収や検体識別番号付けなど、運用面の対応も考慮する。
6. 技術的要件:WGSデプス、RNA seq範囲
7. QC方法、タイミング:標準手法によるシークエンスの場合
受託企業:ヒトゲノムマッピング前のデータを用いて質・量の評価を行い、基準値を満たすデータを取得する。
解析・データセンター:ヒトゲノムマッピング後のデータを用い、質・量の多面的評価を行う。
機関
QCタイミング
WGS
受託企業
解析・データセンター
ヒトゲノム配列へのマッピング前に行う
ヒトゲノム配列へのマッピング後に行う
項目
基準値*
項目
QV30/20以上の塩基割合
75%/90%以上
-
重複リード除去後の塩基数
N: 90G塩基以上
T: 360G塩基以上
-
-
-
マッピング率
-
-
重複率
-
-
インサート長
-
-
読み取り深度
-
-
他者ゲノムの混入
リード数
2,000万リード以上**
-
RNAseq
方針
・中央モニタリングに用いるとともに、各サンプル
ごとの値を研究者及び受託企業に返却
・がん種や試料の種類、ライブラリー作成法、
受託企業等の条件別に集計***
→次年度以降のデータ追加取得等の方針検討に利用
アライメント率
RIN値
参考情報として収集
-
-
*試料の制限により、標準手法での委託でない際には、それに準じたQC基準を定める。
なお、当該基準値を超えたデータ取得を各研究班の予算内で行うことは可能である。
**ポリA精製ライブラリー調整を標準手法とし、その下限を示す。
また、rRNA枯渇処理ライブラリー調整の際は、上記に見合うmRNA由来リードデータ量の取得を目標値とする。
***厚労科研「がん全ゲノム解析等の推進に向けた患者還元、解析・データセンター、ELSI 等に係る技術評価、体制構築についての研究」
班において、各受託企業のシークエンス精度や、当該集計値及びマッピング前のQC値を用いた精度把握を行う。
10