よむ、つかう、まなぶ。
資料3 全ゲノム解析等に係る令和2年から令和4年度までのAMED研究報告 (20 ページ)
出典
| 公開元URL | https://www.mhlw.go.jp/stf/newpage_34345.html |
| 出典情報 | 厚生科学審議会 科学技術部会 全ゲノム解析等の推進に関する専門委員会(第16回 7/226)《厚生労働省》 |
ページ画像
ダウンロードした画像を利用する際は「出典情報」を明記してください。
低解像度画像をダウンロード
プレーンテキスト
資料テキストはコンピュータによる自動処理で生成されており、完全に資料と一致しない場合があります。
テキストをコピーしてご利用いただく際は資料と付け合わせてご確認ください。
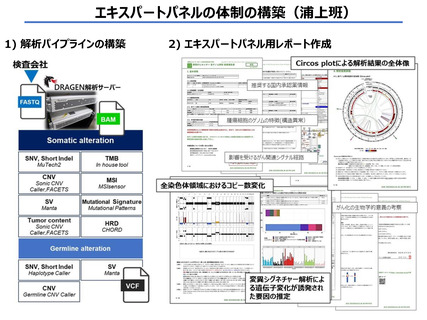
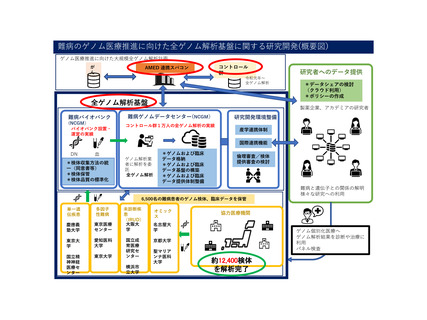
ゲノム解析・クラウド基盤・監視システムの構築(1/2)
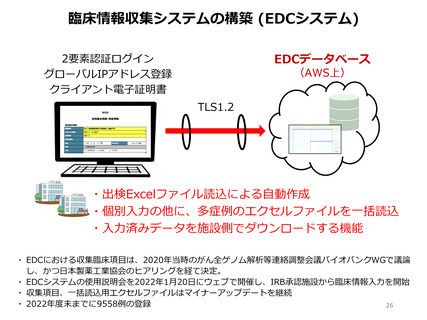
• 研究班、シークエンス受託会社から受け取るゲノムデータのファイル構成、
ロジスティクスを構築
• 受託会社8社からデータを受領
→ HDDを使った場合の暗号化方式の統一は課題
• 統一パイプラインの構築
• SNV, Indel, CNV, SVなど多様なゲノム変異を
共通のツールで同定する解析パイプライン
• QC, アノテーションを含む
• 高度化、高精度化の検討を実施
• 年間1万症例以上のWGS(N: 30x, T: 120x 以上)を解析可能なオンプレミス
のデータ解析環境を構築
• 統一パイプラインを用いて WGS, RNAseq の一次解析を実施
• WGS 12,024症例
• RNAseq 9,317症例
• 解析データを含めると12ペタバイト以上のデータ。Fastqデータだけで4.5ペタ
バイトほどとなる → データ保存領域の確保が課題
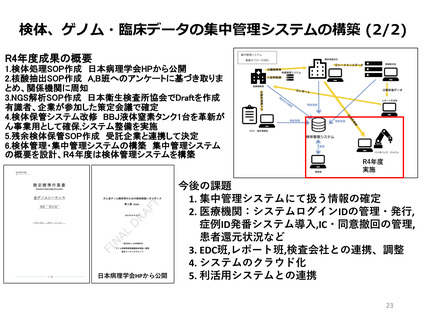
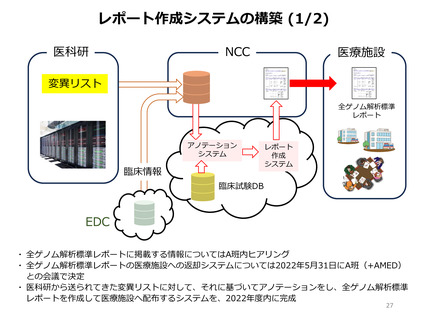
• 解析データを患者還元班(A班、B班)と共有
• 二次解析の環境を提供 → 規模の拡大が課題
20
• 研究班、シークエンス受託会社から受け取るゲノムデータのファイル構成、
ロジスティクスを構築
• 受託会社8社からデータを受領
→ HDDを使った場合の暗号化方式の統一は課題
• 統一パイプラインの構築
• SNV, Indel, CNV, SVなど多様なゲノム変異を
共通のツールで同定する解析パイプライン
• QC, アノテーションを含む
• 高度化、高精度化の検討を実施
• 年間1万症例以上のWGS(N: 30x, T: 120x 以上)を解析可能なオンプレミス
のデータ解析環境を構築
• 統一パイプラインを用いて WGS, RNAseq の一次解析を実施
• WGS 12,024症例
• RNAseq 9,317症例
• 解析データを含めると12ペタバイト以上のデータ。Fastqデータだけで4.5ペタ
バイトほどとなる → データ保存領域の確保が課題
• 解析データを患者還元班(A班、B班)と共有
• 二次解析の環境を提供 → 規模の拡大が課題
20