よむ、つかう、まなぶ。
資料3_全ゲノム解析等に係るAMED研究班からの報告 (31 ページ)
出典
| 公開元URL | https://www.mhlw.go.jp/stf/newpage_38751.html |
| 出典情報 | 厚生科学審議会 科学技術部会全ゲノム解析等の推進に関する専門委員会(第20回 3/18)《厚生労働省》 |
ページ画像
ダウンロードした画像を利用する際は「出典情報」を明記してください。
低解像度画像をダウンロード
プレーンテキスト
資料テキストはコンピュータによる自動処理で生成されており、完全に資料と一致しない場合があります。
テキストをコピーしてご利用いただく際は資料と付け合わせてご確認ください。
• 統一パイプラインのクラウドでの性能検証
• 統一パイプラインのクラウド化に向けて、性能、コスト面から方式や構成ごとの
比較検証を実施
• クラウドの基礎的開発や研究ではなく、全ゲノム解析事業実施にあたって有効な
現行のクラウド技術利用に関する、留意事項や課題の導出を目的とした
• AWS、Google Cloud、Azureにて検証
• AWS ParallelCluster、Batch (AWS, GCP, Azure)、Kubernetes基盤の適用について検証
• 最終報告書を作成
• クラウドでの40症例/Dayの解析性能検証
• オンプレミスで行っている解析と同規模の解析時におけるボトルネック、
コスト最適化を検討(実施中)
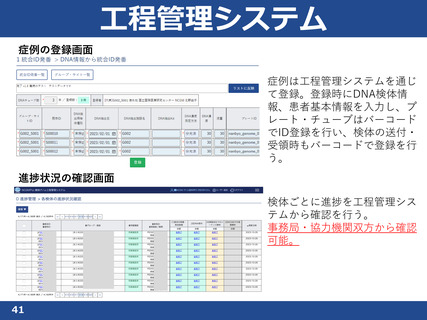
• シークエンス受託会社から受け取るゲノムデータのファイル構成、
ロジスティクスを整理
• 受託会社ごとのロジスティクスのまとめ
•
データ授受についての問題点を各経路で整理
• 標準化に向け情報を整理し、準備室に報告
• 集中管理チームと協力し、シークエンス解析の
オーダーシートの共通化に取組中
31
• 統一パイプラインのクラウド化に向けて、性能、コスト面から方式や構成ごとの
比較検証を実施
• クラウドの基礎的開発や研究ではなく、全ゲノム解析事業実施にあたって有効な
現行のクラウド技術利用に関する、留意事項や課題の導出を目的とした
• AWS、Google Cloud、Azureにて検証
• AWS ParallelCluster、Batch (AWS, GCP, Azure)、Kubernetes基盤の適用について検証
• 最終報告書を作成
• クラウドでの40症例/Dayの解析性能検証
• オンプレミスで行っている解析と同規模の解析時におけるボトルネック、
コスト最適化を検討(実施中)
• シークエンス受託会社から受け取るゲノムデータのファイル構成、
ロジスティクスを整理
• 受託会社ごとのロジスティクスのまとめ
•
データ授受についての問題点を各経路で整理
• 標準化に向け情報を整理し、準備室に報告
• 集中管理チームと協力し、シークエンス解析の
オーダーシートの共通化に取組中
31